蛋白质纯化
来源:MATER METHODS 2012;2:134 | 分类:技术专题 | 翻译:聚生物 | 2019-10-31
Caroline Ritchie (Caroline dot ritchie103, gmail . com),美国爱荷华州立大学
DOI: //dx.doi.org/10.13070/mm.en.2.134
Date:last modified : 2019-10-23; original version : 2012-11-17
Cite as: MATER METHODS 2012;2:134
摘要:对蛋白质纯化柱色谱技术的研究进展进行了综述。
纯化蛋白是许多实验应用所必需的,包括蛋白结构研究和体外生化分析。蛋白质可以从组织中获得,或者更多的是通过它们在模型生物体中的过度表达获取,如培养的细菌、酵母或哺乳动物细胞。蛋白质纯化包括根据蛋白质物理性质的差异,从来源中分离蛋白质。蛋白质纯化方案的目的是用最少的污染物保留最大量的功能蛋白。必须优化蛋白质的纯化方案,以便在最少的步骤内完成这一过程。

图1 Tris缓冲溶液含有Tris碱和它的共轭酸。Tris在25℃时的pKa为8.06,这说明在pH = 8.06时,50%的Tris被质子化(呈酸性),50%被去质子化(呈碱性)。
本文综述了蛋白质纯化中常用的四种柱层析方法,并讨论了它们的优缺点和可能存在的问题。本文还报道了Labome对98种出版物的调查。调查表明,亲和柱层析法是文献中引用的主要方法,主要基于HIS、GST、FLAG tags、size exclusion等。GE医疗是用于蛋白质纯化的试剂和仪器的主要供应商。
开发蛋白质纯化方案
在开发蛋白质纯化方案时,最重要的考虑因素是纯化后的蛋白质的下游应用。蛋白质的数量和纯度必须足以进行实验分析。此外,必须考虑有关蛋白质功能信息,因为折叠良好和功能完整的蛋白是下游研究所需。在纯化和随后的贮藏过程中,可能会发生许多影响蛋白质质量的过程:蛋白质的展开、聚集、降解和功能丧失。在最稳定的条件下,对蛋白质进行尽可能快的纯化,将使纯化方案的能够最大化的成功。
缓冲系统
蛋白质在纯化过程中每一步的溶解条件对维持蛋白质的稳定性和功能至关重要。蛋白质应该被保存在一个缓冲良好的环境中,以防止pH值的突然变化不可逆转地影响其折叠、溶解性和功能。
缓冲液是含有共轭酸碱对的溶液。缓冲液的p H值范围基于其pKa,定义为50%的分子呈酸性,50%的分子呈碱性(图1)。有关缓冲液的一般规则是,缓冲液的pH值应在1.0ph单位内,以提供适当的缓冲容量。这确保了在H+或OH-流入的情况下,有足够数量的酸性和碱性分子中和溶液。因此,缓冲液可以防止pH值的变化,从而对蛋白质的稳定性产生负面影响。

图2. Aktaprime plus系统用于蛋白质的自动色谱分离。图片来源:GE。
一个好的缓冲器必须具备以下特点:
1.水溶性
2.化学稳定性
3.在所需的pH值具备高缓冲能力
4.与分析和实验应用的兼容性
5.与其他解决方案的兼容性

图3 Ni-NTA配体共价连接到交联的琼脂糖基质上,用于选择性纯化多组氨酸标记的蛋白质。组氨酸残基可以通过取代结合水分子(用红色箭头表示)与Ni2+离子配位。然后,咪唑或游离组氨酸可以通过与Ni2+离子配合和取代结合蛋白的能力来洗脱蛋白。图片来源:BioKe。
许多成分可以作为生物缓冲液。最常用的缓冲成分具有接近中性的pKa,因为它们可以在生理pH值下使用。表1列出了四种最常用的生物缓冲液,以及它们可以使用的pH值范围,以及可能影响它们在蛋白质纯化中使用的优缺点。通常,这些缓冲液的浓度高于mM,以确保足够的缓冲能力。
表1 最常用的蛋白纯化生物缓冲液。缓冲液在特定的pH值范围内保持缓冲能力,某些缓冲成分的特性可能会干扰特定的色谱程序或分析。总结自Promega, millipoma, Applichem, Embl.de。
| Buffer | pH range | Advantages and Disadvantages |
| Phosphate | 5.8-8.0 | pH is not dependent on temperature Inexpensive Transparent in the UV range Cannot be used with divalent cations Some proteins may precipitate with sodium phosphate buffer, potassium phosphate buffer can be used. |
| MOPS | 6.5-7.9 | Cannot be autoclaved pH is somewhat dependent on temperature High buffering capacity at physiological pH |
| HEPES | 6.8-8.2 | Cannot be autoclaved pH is somewhat dependent on temperature Can form radicals under certain conditions [2] |
| Tris | 7.5-9.0 | pH is dependent on temperature and dilution Inexpensive Can interfere with the activity of some enzymes [3], [4] Transparent in the UV range |
纯化溶液添加物
除了适当的缓冲系统外,在蛋白质从裂解到储存的纯化过程中使用的溶液通常含有许多其他成分,这些成分在促进蛋白质纯度、稳定性和功能方面起着作用。
蛋白酶抑制剂常被添加到裂解缓冲液和纯化方案的早期步骤中,以防止内源性蛋白酶降解目标蛋白。这些通常在纯化的后期不需要,因为大多数或所有的污染蛋白酶已经从相关蛋白中分离出来。金属螯合剂,如EDTA或EGTA,常被添加到存储缓冲液中。这些金属螯合剂与Mg2+结合,从而通过污染金属蛋白酶防止纯化蛋白的裂解。其他添加剂常用于保护蛋白质不受损害和增强其溶解性。
表2 常用于蛋白质纯化缓冲液中的添加剂,以增加蛋白质的稳定性。总结自Thermo Fisher Pierce和EMBL.de。
| Type | Function | Commonly Used Reagents |
| Reducing Agents | Protect against oxidative damage | 2-mercaptoethanol (BME) Dithiothreitol (DTT) Tris (2-carboxyethyl) phosphine (TCEP) |
| Protease Inhibitors | Inhibit endogenous proteases from degrading proteins | Leupeptin (serine and cysteine protease inhibitor) Pepstatin A (aspartic acid protease inhibitor) PMSF (serine protease inhibitor) |
| Metal Chelators | Inactivate metalloproteases | EDTA EGTA |
| Osmolytes | Stabilize protein structure and enhance solubility | Glycerol Detergents (e.g., CHAPS, NP-40, Triton X-100) Sugars (e.g., glucose, sucrose) |
| Ionic Stabilizers | Enhance solubility | Salts (e.g., NaCl, KCl, (NH4)2SO4 |
只有在必要时才应使用添加剂。通常需要反复试验来确定有利于特定蛋白质纯化方案时使用添加剂。
其他注意事项
在纯化过程中,其他因素也有助于蛋白质的稳定性。在蛋白质纯化过程中,最少的操作总是最好的。设计一个在最短时间内使用最少步骤的纯化方案,确保功能蛋白的最高产量。此外,在整个纯化过程中保持蛋白质的低温通常是最好的。通常,纯化是在4℃下进行的,因为较低的温度既减慢了蛋白质分解的速度(在有污染蛋白酶的情况下),又促进了蛋白质的结构完整性。
表达系统对蛋白质纯化的影响
在纯化感兴趣的蛋白质之前,必须制备初始的粗样品。首先要考虑的是所需蛋白质的来源,这在实际纯化之前就已经发生了。这可能是一种天然来源,如肝脏、肌肉或脑组织,但在后基因组领域,研究人员从天然来源纯化蛋白质的情况相对较少。然而,如果研究者希望将催化活性与特定蛋白质序列联系起来,有时仍有必要的。
如今,从重组源中纯化蛋白质的情况要普遍得多。需要提前做出重要决定,以优化后续净化。研究者需要考虑蛋白质的最终用途(如酶分析、结构研究、抗体生成),因为这将决定最终蛋白质制备所需的数量和纯度。一个重要的考虑因素是表达系统的选择(见Labome文章重组蛋白表达:载体宿主系统)以讨论基于文献的无偏调查的最广泛使用的表达系统。虽然对蛋白质表达的详细讨论超出了本文的范围,但在这一点上有几个基本点值得考虑,因为它们直接影响到蛋白质的后续纯化。
哪种表达系统能提供最高的表达水平?一般来说,表达水平越高,就越容易获得大量高纯度的蛋白质。
如果选择大肠杆菌表达,最终目的是可溶性表达还是不溶性的包涵体的形式表达?分离包涵体本身可以构成一个重要的纯化步骤,由于重组蛋白在包涵体中的丰度较高,分离包涵体本身就是一个重要的纯化步骤;这必须与从包涵体中溶解并随后重新折叠目标蛋白的难易程度和可溶性蛋白的最终产量相平衡[5]。为了优化包涵体的功能蛋白产量,人们做了大量的工作[6]。
另一个需要考虑是是否针对细胞内或细胞外(分泌)表达。细胞内表达需要从大量的宿主细胞蛋白中纯化该蛋白。相反,目标蛋白的有效分泌会导致从少量分泌的宿主蛋白中纯化出蛋白,特别是在无血清条件下生长的宿主细胞[7]。
样品制备
无论目标蛋白的来源是什么,作为纯化起始点的粗样的初始制备都是重要的,需要在考虑表达和纯化策略的同时加以考虑。
目的蛋白的胞外分泌可能允许使用一种快速而直接的亲和纯化方案[7];唯一需要的样品制备可能是将条件培养基从贴壁细胞中倒出或低速离心去除悬浮细胞。当然,可能有必要添加蛋白酶抑制剂或调整pH值为第一步色谱准备,但这些都是简单的步骤。然而,如果要进行离子交换作为第一个净化步骤,样品可能首先需要脱盐。如果要处理大量的条件培养基,这在技术上是有挑战性的;根据需要脱盐的介质的体积,通常采用透析或交叉流过滤。
如果靶蛋白在细胞内表达,则首先需要通过离心分离获得细胞,然后再将其重新悬浮在适当的裂解缓冲液中。如上所述,裂解缓冲液需要包含适当的缓冲液和其他添加剂,以确保目标蛋白的最大稳定性。如果研究者要避免在柱层析之前进行耗时的缓冲液交换步骤,则样品/裂解缓冲液的成分也需要与随后的纯化步骤相兼容。
接下来,需要一种有效的方法来溶解细胞。文献中描述了各种方法。大肠杆菌细胞可以用法国压力裂解法(虽然这种方法不容易扩展)、超声波裂解法或以洗涤剂为基础的裂解法(市面上有许多裂解试剂)裂解。在裂解液中加入溶菌酶可以提高洗涤剂裂解的效率。以清洁剂为基础的裂解反应非常温和,不会导致细菌DNA的显著剪切。因此,通常需要加入DNAase制剂(高纯度的制剂在市场上可以买到)来降低样品的粘度,制备出具有良好流动特性的样品[8]。
超声波或洗涤剂为基础的方法也可以溶解哺乳动物和昆虫细胞。如果核膜明显溶解,可能需要DNAase处理来降低样品粘度[8]。
一旦细胞(微生物/昆虫/哺乳动物)被溶解,通常需要通过离心法去除细胞碎片(通常在4℃下15 000 x g持续15分钟,以避免色谱柱堵塞[8]。得到的上清液是柱层析纯化目标蛋白的起点[8]。
柱层析简介
柱层析设备
柱层析法的原理是将一个大的蛋白质池分离成许多小的蛋白质池,其中一些富含感兴趣的蛋白质。虽然柱层析设备昂贵且专业,但纯化只需要基本设备。

图4.使用蛋白质A、G或L.A.的亲和纯化示意图。抗体包含几个感兴趣的区域:对同一类的所有蛋白质相同的Fc(片段,常数)、对每种抗体提供特异性的Fv(片段,变量)和实际接触特异性抗原的Fab(片段,抗原结合)。b.特定种类的抗体可通过亲和纯化进行非选择性纯化,其中配体(蛋白质A、G或L)与基质结合。蛋白A、G或L也可用于纯化特定蛋白。在这种情况下,抗体将作为中间配体,为其抗原提供选择性。
柱层析基本设备:
1.固定相:一种惰性基质,通常带有一个附加的官能团以促进蛋白质相互作用,用于分离蛋白质。固定相和官能团的选择取决于所用色谱的类型和所用的方法。
2.圆柱:一个圆柱形的玻璃容器,有不同的长度和直径供选择。可以购买带有固定相的预包装柱,并准备连接到自动色谱系统(在本节第2部分中讨论),也可以购买空的手动包装柱。自动流动色谱法与重力流动色谱法需要不同类型的色谱柱。
3.溶剂:含有添加剂的缓冲液,用于平衡、洗涤和洗脱固定相中的蛋白质。不同类型的色谱需要不同的溶剂条件。
4.收集管:用于收集洗脱样品的容器。自动分馏收集器需要特殊的管;但是,任何管或容器都适合手动分馏收集。
5.纯度测定:测定样品中特定蛋白质与总蛋白质相对含量的方法。在进行纯化方案的下一步之前,必须在每一步之后测定含有感兴趣蛋白质的组分。一些分析纯度的常用方法将在后面的章节中讨论。
柱层析系统
柱层析可以用自动化系统进行(图2),该系统使用泵迫使溶剂以设定的流速通过填充柱,也可以通过重力流来运行。自动化和重力流系统都可以与自动分馏收集系统耦合。每个系统都有优点和缺点。GE Healthcare AKTA FPLC系统是非常常见的选择[9-18]。
表3 蛋白质的色谱分离系统。资料来源:GE。
| Type of System | Advantages | Disadvantages |
| Automated | Can let run by itself Often coupled to an absorbance detector Can program equilibration and wash steps Easy to set up a gradient for elution Very reproducible |
Requires specific, costly equipment The maximum flow rate is dependent on the pressure limit of the column |
| Gravity Flow | Less expensive The user has more control Can make adjustments during run |
More labor intensive The flow rate is limited by gravity |
柱层析类型
四种主要的柱层析法包括亲和层析法、离子交换层析法(IEX)、疏水相互作用层析法(HIC)和尺寸排阻层析法(SEC)。大多数净化方案要求使用以上一种以上的色谱程序,以获得下游应用所需的纯度。选择最合适的色谱方法和这些方法的顺序是优化蛋白质纯化方案的关键。
表4 四种最常见的用于蛋白质纯化的柱层析方法。
| Type of Chromatography | Separates Proteins By | Bind With | Elute With |
| Affinity | A specific interaction | No competing ligand | Competing ligand (specific); conditions that disrupt protein/protein interactions (non-specific) |
| Ion Exchange | Net surface charge | Low ionic strength | High ionic strength; Increased (cation exchange) or decreased (anion exchange) pH |
| Hydrophobic Interaction | Hydrophobicity | High ionic strength | Low ionic strength |
| Size Exclusion | Hydrodynamic radii |
通过分析蛋白质的序列,可以识别出可能有助于其纯化的独特特征。蛋白质的大小和电荷(在特定的pH值下)可以确定,同时还可以确定大量的疏水基团。
亲和层析
亲和层析依赖于蛋白质与基质结合配体的特异性可逆结合。配体可以直接与感兴趣的蛋白质结合,也可以与共价附着在蛋白质上的标签结合。亲和层析法通常是最有效的纯化方法,通常用于纯化方案的早期阶段。根据下游的应用,亲和纯化可能是获得足够纯度所需的唯一层析步骤。

图5 蛋白质上的净电荷受其溶剂的pH值的影响。在pH=pI时,蛋白质的净电荷为零,因此不会与阳离子交换或阴离子交换固定相结合。调节pH值,使其高于或低于pI,就会产生净电荷,并使蛋白质与阴离子交换(pH > pI)或阳离子交换(pH < pI)固定相结合。
亲和层析的固定相是由一个与配体共价连接的惰性基质构成的,配体特异性地与蛋白质或蛋白质组结合。惰性基体通常由交联的琼脂糖或聚丙烯酰胺组成。蛋白可通过亲和层析进行选择性或非选择性纯化。在选择性亲和层析中,使用特定于蛋白质或共价附着标签的配体。在非选择性亲和层析中,如蛋白A、G、L用于免疫球蛋白,或肝素用于DNA结合蛋白,或凝集素用于糖蛋白,配体与一组具有类似结合能力的蛋白结合。
在两种亲和层析中,蛋白质都是在影响蛋白质(或标签)与其配体结合的条件下装载在色谱柱上的。结合蛋白在不破坏特异性相互作用的条件下被洗涤,但这种条件可以破坏污染蛋白与固定相之间的任何非特异性相互作用。然后用含有竞争性分子或破坏所有蛋白质/蛋白质相互作用的条件的缓冲液洗脱结合蛋白。相互竞争的分子与配体结合,取代感兴趣的蛋白质。这种竞争性分子通常通过另一种色谱方法或透析从相关蛋白中去除。通过干扰所有蛋白质/蛋白质相互作用来从固定相中洗脱蛋白质的方法包括调整缓冲液的pH值或离子强度。这些方法会影响蛋白质的稳定性,建议将洗脱的蛋白质立即中和或稀释,以减少蛋白质的损伤。对于一些亲和层析的形式,已经描述了替代洗脱条件,以最大限度地提高功能蛋白的产量[19,20]。
表5 用于特异性和典型洗脱条件的具有官能团(配体)的选择性和非选择性亲和层析形式的示例。总结自Thermo Fisher Pierce和GE。根据Labome对200多种出版物的调查,不同类型栏目的常见供应商见下文。
| Protein to Purify | Ligand | Elute With | ||
| Antibody (antigen-specific) | Antigenic peptide | Free peptide | ||
| Polyhistidine-tagged protein | Ni2+ or Co2+ | Imidazole or free histidine | ||
| FLAG-tagged protein | FLAG-specific antibody | FLAG peptide or low pH | ||
| GST-tagged protein | Reduced glutathione | Free glutathione | ||
| Myc-tagged protein | Myc-specific antibody | Low pH | ||
| Antibody (class-specific) | Protein A , G, or L or protamine | Extremes in pH | ||
| DNA-binding protein | Heparin | High ionic strength | ||
在设计用于蛋白表达的质粒时,亲和标记可以插入N或c端(或在罕见的情况下,插入具有已知结构的柔性蛋白环中),以帮助纯化。有关常见标签和标签相关讨论的列表,请参见Labome关于蛋白质/protide标签的调查。
抗体通常是根据抗体和抗原之间发生的高度特异性相互作用(抗体识别的序列)来纯化的。含有抗原的肽可偶联到基质上特异性结合抗体。降低洗脱缓冲液的pH值,干扰抗体/肽相互作用,释放结合抗体。这种方法通常用于从粗血清中分离抗体。

图6. 离子交换色谱法。蛋白质以低离子强度与带电的固定相结合。结合蛋白可以通过增加缓冲液的离子强度或通过调节pH值来洗脱。
蛋白质也可以通过非选择性的方式亲和纯化。在非选择性纯化中,固定相上的配体与一组具有相似结合伙伴的蛋白质结合。非选择性亲和纯化的一个例子是dna结合蛋白的纯化。肝素在结构和电荷上都与DNA相似,可作为DNA结合蛋白亲和纯化的配体。虽然所有的dna结合蛋白理论上都可以与这个固定相结合,但是大多数其他的蛋白在没有结合的情况下就会通过,从而使我们感兴趣的蛋白质得到充分的富集。另一个例子是通过将其恒定(Fc)区域与配体、蛋白A、G或l结合来富集抗体。GE Healthcare蛋白A亲和柱是常见的选择[21-23],蛋白G也是如此[24]。对于IgM,鱼精蛋白亲和层析可以使用[25]。
使用类似的结合模式(抗体与蛋白A、G或L配体结合),抗体的抗原结合(Fab)区域仍可与特定抗原结合。因此,可以根据偶联的配体/抗体和抗体/抗原之间的特异性相互作用来纯化特定的蛋白质。
常见问题及排除方法见表6。
表6. 亲和层析故障排除。来自:GE。
| Problem | Cause | Solution |
| Protein does not bind | Tag was not translated or is not accessible | Check plasmid sequence or move the tag to a different location |
| Binding conditions are not appropriate | Adjust buffer conditions | |
| Not enough time was allowed for binding | Decrease flow rate or stop column to allow incubation | |
| Protein does not elute | Affinity between ligand and tag is very high | Increase concentration of competitor (specific) or stringency of conditions (non-specific) |
| Protein aggregated on column | Adjust buffer conditions for more protein stability | |
| Low resolution | Flow rate is either too fast or too slow | Adjust flow rate |
| The column was not washed sufficiently | Wash with higher stringency buffer; Clean stationary phase according to manufacturer | |
| Protein aggregated on column | Adjust buffer conditions for more protein stability | |
| Elution conditions are not stringent enough | Increase concentration of competitor (specific) or adjust conditions (non-specific) | |
| Protein loses activity during procedure | Protein is unfolded or aggregated | Adjust buffer conditions for more protein stability |
| A cofactor required for activity was removed during purification | Add cofactor |
离子交换色谱法
离子交换色谱(IEX)通过蛋白质和带电固定相之间的静电相互作用,根据蛋白质的净表面电荷来分离蛋白质。IEX存在两种类型:(1)阴离子交换(与负电荷蛋白结合的带正电荷的固定相);(2)阳离子交换(与带正电荷的蛋白质结合的带负电荷的固定相)。离子交换层析是蛋白质纯化方案中常用的中间步骤,但在纯化过程中较早或较晚使用时,可对某些蛋白质产生较高的分辨力。
所有的蛋白质都表现出一种净电荷,这种净电荷依赖于蛋白质的氨基酸组成和任何共价连接的修饰。当溶剂与蛋白质交换氢离子时,蛋白质的净电荷受溶解于其中的溶剂的pH值的影响。蛋白质的等电点(pI)是蛋白质不带净电荷时的pH值。当pH值高于pI时,蛋白质带净负电荷,而pH值低于pI时,蛋白质带净正电荷。因此,可以调整溶剂的pH值以促进与IEX的结合或促进结合蛋白的洗脱。
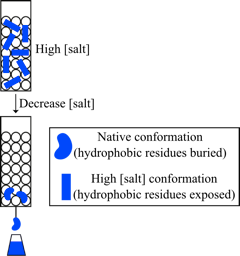
图7 疏水作用色谱法。在高离子强度下,蛋白质部分脱溶,导致它们采用交替构象,其中通常埋藏的疏水性残基暴露得更多。这些残基可以与共轭于基质的疏水官能团形成疏水相互作用。降低离子强度会使蛋白质重新形成其固有的构象,从而掩盖其疏水性残基。这减少了蛋白质和固定相之间的疏水相互作用,促进了蛋白质的洗脱。
理论上,如果相应地调节缓冲液的pH,则所有蛋白质均可与阳离子交换和阴离子交换结合。然而,对于蛋白质纯化而言,蛋白质的稳定性是选择纯化条件的最重要考虑因素,因此,蛋白质结合柱是最合适的。因此,有必要确定结合任何形式的离子交换层析所需的条件是否影响蛋白质的稳定性和功能。典型地,结合一种类型的离子交换的条件比另一种类型更适合特定的蛋白质。
了解蛋白质的等电点可以帮助确定最合适的离子交换色谱类型。在线工具可用于计算蛋白质的理论PI。这些计算完全基于蛋白质的氨基酸序列,不考虑蛋白质的三维结构。在其固有状态下,蛋白质的某些残基比另一些蛋白质更容易暴露,因此,实际的PI和净表面电荷有时与理论上确定的不同[26]。氨基酸的相对分布也会影响蛋白质的PI[27],在理论PI计算中没有考虑到这一点。有些技术允许实验测定蛋白质的自然状态下的PI,包括电泳等电聚焦[28],毛细管等电聚焦[29],以及最近的一种基于高通量发光的方法[30]。
通常,蛋白质与IEX的结合必须通过反复试验来确定,使用具有一定pH值范围的溶剂,以确定蛋白质保留的最佳pH值。与pI相距约一个pH单位的溶剂通常足以与蛋白质结合[31];然而,在某些情况下,需要与pI相距更远的pH值[32]。
IEX的固定相由具有共价键的惰性琼脂糖基或聚合物基质组成。基质颗粒有多种尺寸,既可以是非多孔的,也可以是含有可变尺寸的孔隙。基质的选择取决于所需的结合容量、分辨率和流量。较小的颗粒尺寸提供更高的分辨率,但需要较低的流速和运行时间。多孔基质比非多孔基质具有更高的结合能力.在非多孔基质中,蛋白质不能进入树脂,因此,比多孔基质提供更高的样品回收率、更高的分辨率和更短的运行时间。一项研究表明,对于大多数蛋白质来说,当被多孔基质和非多孔基质分离时,分辨率和恢复是相似的;然而,对于较大的蛋白质,多孔基质会导致基于尺寸排除效应的分辨率下降[33]。

图8.以盐(硫酸铵)梯度递减的疏水作用柱洗脱的蛋白质。对所研究蛋白质的总蛋白含量和活性进行了分析。从蛋白质活性可以看出,以45分为中心的峰值包含了我们感兴趣的蛋白质。从http://www.insectscience.org/9.04/。
IEX中最常用的四个荷电官能团如下图所示。它们分为强交换器和弱交换器,强交换器比弱交换器的pH值范围更大(图8)。
阴离子交换(正电荷固定相):
1.季铵(Q)强阴离子交换器
2.二乙基氨基乙(DEAE) -弱阴离子交换器
阳离子交换(负电荷固定相):
1.甲基磺酸盐(S) -强阳离子交换剂
2.羧甲基(CM) -弱阳离子交换器
当结合所需的pH值是非常酸性或碱性时(假设该pH值也适合于保持蛋白质的稳定性),应使用强交换剂,因为官能团在较大的ph范围内保持电荷[34]。研究表明,弱交换剂能够减少某些蛋白质的保留,使其在较低离子强度下洗脱[35]。由于高离子强度会影响某些蛋白质的稳定性[36],弱交换剂可能更适合于不需要极端pH值来结合的蛋白质。
在离子交换色谱中,固定相首先用低离子强度缓冲液平衡。然后将蛋白样品装入用于平衡的相同的低离子强度缓冲液中的固定相。结合蛋白在用高离子强度缓冲液或在某些情况下用pH值改变的缓冲液洗脱前要进行大量清洗(图6)。洗脱缓冲液中的反离子与带电的固定相相互作用,取代结合蛋白。某些盐类在离子交换色谱中比其他盐类更有效,因为它们可以取代结合蛋白并对蛋白稳定性[37]产生影响。通常用NaCl或KCl洗脱,Na+或K+作为阳离子交换层析的对位离子,Cl-作为阴离子交换层析的对位离子。或者,可以改变缓冲液的pH值,以减少蛋白质的电荷并破坏其与固定相的相互作用。对于与阳离子交换固定相结合的蛋白质,增加缓冲液的pH值会使蛋白质上的正电荷减少,从而从柱中洗脱出来。对于结合在阴离子交换固定相上的蛋白质,降低缓冲液的pH值会导致蛋白质上的负电荷减少,从而从色谱柱中洗脱出来。
或者,可以调节缓冲液的pH,使得所关注的蛋白质不与离子交换固定相结合,同时污染蛋白质。在这种情况下,感兴趣的蛋白质被收集在流通相中,而一些污染的蛋白质通过它们与固定相的结合而被去除。
与其他色谱方法一样,离子交换色谱可能需要一些故障排除,以确定蛋白质结合、蛋白质洗脱和足够分辨率的最佳条件。
表7.离子交换色谱故障排除,来自:GE.
| Problem | Cause | Solution |
| Protein does not bind | Column was not equilibrated | Run more equilibration buffer through the column and reload protein |
| The ionic strength of binding buffer is too high | Lower ionic strength of the buffer | |
| pH is not far enough from pI | Adjust buffer pH (lower for cation exchange, higher for anion exchange) | |
| Protein does not elute | Ionic strength of elution buffer is too low | Increase ionic strength |
| Protein aggregated on column | Adjust buffer conditions for more protein stability | |
| Low resolution | Flow rate is either too fast or too slow | Adjust flow rate |
| The column was not washed sufficiently | Wash with a higher ionic strength buffer; Clean stationary phase according to manufacturer | |
| Protein aggregated on column | Adjust buffer conditions for more protein stability | |
| Protein loses activity during procedure | Protein is unfolded or aggregated | Adjust buffer conditions for more protein stability |
| A cofactor required for activity was removed during purification | Add cofactor |
疏水相互作用色谱法
疏水相互作用层析(HIC)基于它们的疏水性分离蛋白质,并且通常用作纯化方案中的中间步骤。蛋白质结合在高离子强度缓冲液中的固定相,因此,HIC通常可以在离子交换层析之后立即进行,不需要缓冲液交换或稀释。HIC也通常在氨硫酸盐沉淀之后进行,该过程可用于通过快速而不是全部的蛋白质沉淀蛋白质来除去蛋白质。HIC有时可应用于纯化方案的早期步骤或作为从感兴趣的蛋白质中除去痕量杂质的最后步骤。
溶液中盐离子的存在会导致蛋白质的部分展开和通常埋藏的疏水残基的暴露。与固定相结合的蛋白质采用其天然构象作为缓冲液,离子强度较低。这减少了与固定相相互作用的疏水残基的暴露,有助于从柱中洗脱。对于通过降低离子强度而自发复性的蛋白质,HIC是一种有价值的色谱纯化方法。
通常情况下,缓冲液的离子强度应尽可能低,以结合相关蛋白,同时防止其沉淀。如果蛋白结合所需的离子强度高,导致相关蛋白沉淀,可以使用较低的离子强度。在这种情况下,色谱程序可以用来分离所有的结合蛋白,从蛋白的兴趣流动没有结合。
在将样品装入柱之前,固定相必须与高离子强度缓冲液(与蛋白质样品所用的缓冲液相同)平衡。然后将样品装入柱中,对柱进行大量清洗,并用低离子强度缓冲液洗脱蛋白质(图7和图8)。
疏水作用色谱中使用的固定相由交联琼脂糖或合成共聚物的基质组成。然后,烷基或芳基配体与碱基质结合,为疏水分子提供特异性分离。
官能团类型:
- 烷基:不同长度的烃链;通常采用丁基或辛基。随着烷基链长度的增加,固定相的结合能力增加[38]。官能团结合完全基于蛋白质的疏水性
- 芳基-由芳香环衍生出的官能团;通常是苯基。芳基基团提供了更多的特异性,因为蛋白质也可以通过碱基堆积与官能团相互作用。
用于结合和洗脱的盐的类型和浓度必须根据每种蛋白质经验确定。此外,必须在洗脱后确保蛋白质的重折叠和功能。
与其他形式的柱层析一样,最优的HIC程序可能需要大量的故障排除。每种蛋白质都需要调整缓冲条件和固定相,以确保最佳分离。
表8 疏水相互作用色谱法的故障排除,来源:GE
| Problem | Cause | Solution |
| Protein does not bind | Column was not equilibrated | Run more equilibration buffer through the column and reload protein |
| The ionic strength of binding buffer is too low | Increase ionic strength of the buffer | |
| Protein aggregated at the ionic strength used | Decrease ionic strength of buffer or try a different salt | |
| Protein does not elute | Ionic strength of elution buffer is too high | Decrease ionic strength |
| Protein aggregated on column | Adjust buffer conditions for more protein stability | |
| Retention is too high | Try a different stationary phase that offers less retention | |
| Low resolution | Flow rate is either too fast or too slow | Adjust flow rate |
| Column was not washed sufficiently | Wash with a lower ionic strength buffer; Clean stationary phase according to manufacturer | |
| Protein aggregated on column | Adjust buffer conditions for more protein stability | |
| Poor retention on column | Try a different stationary phase that offers greater retention | |
| Protein loses activity during procedure | Protein is unfolded or aggregated | Adjust buffer conditions for more protein stability |
| Protein did not return to its native conformation | Try binding with a different salt or at a lower ionic strength; Include additives for protein stability | |
| A cofactor required for activity was removed during purification | Add cofactor |
尺寸排阻层析(SEC)
尺寸排阻层析(Size exclusion chromatography,SEC)根据蛋白质的流体动力学半径分离蛋白质,这种性质由分子的大小和形状决定。与前面描述的其他层析法不同,蛋白质在SEC中不与固定相结合。相反,蛋白质是通过它们在惰性固定相中的导航速度来分离的(图9)。

图9 三种不同流体力学半径的蛋白质的混合物被加载到一个层析柱上。大的蛋白质首先脱溶,因为它们不能进入基质的孔隙,并有一个直接的途径通过柱。更小的蛋白质可以进入孔隙,有一个更复杂的路径,因此,需要更长的时间来穿过基质和从柱中洗脱
Superdex通常是典型SEC程序的最佳选择,因为它与大多数溶剂兼容,并提供所有可用的固定相的最高分辨率。
与其他色谱方法一样,SEC也有优点和缺点,是否决定使用这种方法取决于蛋白质及其下游的应用。
SEC的优点:
- 提供缓冲交换和脱盐。
- 使其他净化技术可能无法分离的相似物种(例如截断和寡聚体)分离。
- 与多种溶剂相容。
4.保留和洗脱不依赖于任何特定的蛋白质性质。
SEC的缺点:
- 性能对柱填料非常敏感。
- 蛋白质与树脂之间存在非特异性的相互作用,从而降低了分辨率。
- 复杂蛋白质混合物分辨率低。
- 样品必须以小体积装载,以获得足够的分辨率。对于高浓度沉淀的蛋白质来说,这可能是个问题。
优化SEC条件以获得最大的分辨率通常是耗时的,但某些因素会对分辨率产生巨大的影响。
SEC提高蛋白质分辨率的方法:
1.尽量减少样本量。体积越小,洗脱部分的扩散越小。
2.在缓冲液中加盐。少量的盐将有助于防止蛋白质和固定相之间的非特异性相互作用。这将使所有的蛋白质分子在色谱柱的长度上保持一致。
3.使用中等流速。过快地运行色谱柱不会让小分子有时间进入基质孔隙,而流速过慢则会让样品扩散有更多的时间。
4.确保样品和溶剂粘度相似。调整采样条件以近似匹配缓冲器。
5.调整柱长。太短的色谱柱不能进行足够的蛋白质分离。或者,柱太长会导致蛋白质样品扩散。
6.重新包装柱子。柱填料对蛋白质的分离有很大的影响。
如果颗粒没有很好地分散,或者如果气泡被困在色谱柱中,分子无法正确地通过固定相。此外,如果允许层析柱干运行,必须重新填料。无法解释的低分辨率,通常要归咎于填料不良。
由于SEC不能根据与官能团的相互作用来分离蛋白质,所以所有的蛋白质都是在相同的条件下洗脱的,因此,只能对水动力半径非常不同的蛋白质获得分辨率。因此,在纯化方案的初始阶段,当有许多污染蛋白质时,SEC不是一个合适的色谱方法。然而,SEC确实提供了一种快速、可靠的方法,用于在净化的早期或中期阶段去除样品中的盐或小分子。在纯化的最后阶段,当仅存在微量污染物时,SEC是一种有价值的蛋白质分离和交换的方法。
蛋白质的洗脱与分析
蛋白质洗脱方法
与固定相结合的蛋白质用破坏结合相互作用的溶剂条件洗脱。这些洗脱条件因层析类型和感兴趣蛋白质的性质而异。蛋白质洗脱有三种常用方法:分批洗脱、分步洗脱和线性梯度洗脱。最佳方法的选择取决于所执行的层析类型和所需的分辨率。
- 分批洗脱法-单个洗脱条件在一个步骤中置换所有结合蛋白。这种机制最适用于基于非常特殊相互作用(即亲和层析)的层析过程。分批洗脱不提供任何分辨率,但它是快速去除污染物的理想方法。这需要预先了解置换感兴趣蛋白质所需的缓冲条件。
- 分步洗脱--连续进行多步洗脱,每一步都有更严格的条件。在逐步洗脱中,所收集的组分数取决于洗脱条件的个数。分步洗脱比分批洗脱具有更好的分辨率,但比线性梯度洗脱的分辨率低。
- 线性梯度洗脱-收集多个连续的组分,同时线性调整洗脱条件。线性梯度为离子交换层析和疏水相互作用层析提供了最高的分辨率。通常,会收集大量连续的组分。
尺寸排阻色谱法不需要这些洗脱方法,因为蛋白质和固定相之间没有相互作用。蛋白质被装载到色谱柱上,在整个过程中不改变缓冲条件的情况下,收集大量组分,直到所有蛋白质被洗脱。
理想情况下,层析柱的洗脱缓冲液与后续层析柱相容,无需在纯化步骤之间进行缓冲液交换或透析。
较不常用的层析法
羟基磷灰石(羟基化磷酸钙)层析法是一种蛋白质纯化技术,最初描述于20世纪50年代中期[42]。球形羟基磷灰石的商业化应用使羟基磷灰石柱层析技术成为一种可行的技术[43]。蛋白质与羟基磷灰石柱相互作用的机理是复杂的。蛋白质最常被磷酸盐梯度洗脱。(讨论情况见[43])。羟基磷灰石与其他色谱技术具有不同的选择性,可以为纯化难纯化的蛋白质或作为最终的抛光步骤提供有用的补充。羟基磷灰石层析已成功地用于治疗级抗体的纯化[44]。
色谱聚焦法是一种基于蛋白质pI的柱色谱分离方法。蛋白质与专门的离子交换介质结合,用pH梯度洗脱。染色质聚焦的选择性不同于其他技术,因此作为最后的“抛光”步骤是有用的[45]。
纯度分析
在每次色谱分离后,必须对各组分进行分析,以确定含有感兴趣蛋白质的组分以及各组分的相对纯度。在每一步之后,这一分析都是必要的,以决定哪些部分应该合并以供以后使用。为了确定纯度,需要一种能够测量特定蛋白质的量相对于总蛋白质量的分析方法。以下分析通常用于纯度分析:
1. 十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)法 -一种根据大小来分离蛋白质的变性凝胶。商业上可用的着色剂可以对样品中的所有蛋白质进行可视化表示,从而对蛋白质纯度进行定性评估(图10)。SDS-PAGE对于馏分的不是理想的高通量分析方法,可能需要几个小时;然而,它是最常用的,因为它容易,便宜,适合任何蛋白质。

图10 蛋白质纯化过程中所收集样品的SDS-PAGE。凝胶被染色以显示所有蛋白质。来源:http://www.omicsonline.org/。
2. 光谱法-分析蛋白质光学性质的方法。这种分析蛋白质纯度的技术只适用于具有独特光谱特征的蛋白质,如细胞色素P450s。这个家族的蛋白质吸收的光的波长是其他蛋白质吸收不到的(大约是420纳米[46]),所以将420纳米的吸收度与280纳米(所有蛋白质吸收的波长)进行比较,可以提供蛋白质纯度的定量测量。该方法快速、高效,但仅适用于部分蛋白质。
3. 蛋白质活性分析法-取决于感兴趣的蛋白质的酶促测试。这种评估蛋白质纯度的方法通常与另一种形式的蛋白质浓度测定相结合,以计算相对于总蛋白质浓度的活性。活性分析仅适用于具有易于以高通量形式监测的活性的蛋白质,例如蛋白酶。对于某些蛋白质,活性测定为蛋白质的检测提供了一种快速、可靠的方法。活性测量通常是酶蛋白检测的理想选择,因为失去活性的蛋白质可以被排除在随后的使用之外。
纯化蛋白的存储
当蛋白质被认为足够纯净可以用于实验研究时,它们应该被适当地储存起来。最终存储缓冲液的选择与纯化过程中使用的缓冲液的选择同样重要,应取决于蛋白质的稳定性和纯化后下游应用所需的条件。通常,在纯化方案中最后一步是选择尺寸排阻层析,因为存储缓冲液可以在这个层析步骤中有效交换缓冲液。纯馏分可汇集起来立即储存。或者,最后的混合馏分可以在存储之前透析到选择的缓冲液中。
蛋白质的贮藏条件取决于目标蛋白质,因此应该对蛋白质进行优化,使目标蛋白质在长时间的贮藏过程中保持结构和功能的稳定性。为了提高蛋白质在贮藏条件下的使用寿命,常常在贮藏缓冲液中添加添加剂,由于每种蛋白质的特性不同,因此常常需要反复试验来确定最佳的贮藏条件。
高通量蛋白质纯化
在这个后基因组时代,蛋白质的高通量纯化无论是用于结构测定还是用于药物发现/化合物筛选,都引起了人们的极大兴趣。为了筛选广泛的并为所需的最终用途选择的最佳结构,研究人员利用了一系列商用自动化/机器人系统,能够快速、平行、半自动化地纯化亲和标记蛋白[47,48]。蛋白质的常规纯化方法仍然适用;液体处理机器人/自动化平台仅用于简化和加速纯化过程。
膜蛋白
细胞产生的蛋白质约20-30%为膜蛋白,50%左右的小分子药物作用于膜蛋白[49]。因此,人们对解决膜蛋白的结构有很大的兴趣。完整膜蛋白纯化的一个关键步骤是在保持其功能完整性的同时从脂质双分子层中溶解。典型的方法是先离心分离胞内膜,然后用去垢剂溶解整体膜蛋白,再用高速离心去除不溶性膜残基[50-52]。大量的洗涤剂被用于膜蛋白增溶[53-55],在没有任何文献或实验室先例的情况下,研究者需要根据经验确定其特定蛋白的最佳洗涤剂。所述可溶膜蛋白可通过柱层析进行纯化,其方法与可溶性蛋白基本相同。然而,纯化缓冲液需要含有洗涤剂,以保持蛋白质在可溶性状态[56,57]。膜蛋白的纯化通常是极具挑战性的,因为在从脂质双层膜上初始去除并通过各种纯化步骤后,蛋白质功能完整性和聚集性会丧失。尽管如此,有几个小组已经成功地纯化了足够数量的膜蛋白用于结构测定(参见结构基因组学联盟)。最近,纳米圆盘已经成功地用于B家族GPCR的亲和纯化(参见Labome关于纳米圆盘的文章)。
Labome综述了有关蛋白质纯化的文献
Labome调查了引用蛋白质纯化方法的出版物。表10列出了主要供应商和净化方法。亲和力和大小排除方法是文献中最常用的方法。GE Healthcare是所有方法的主要供应商,而Qiagen是基于HIS标签的标签供应商,和基于FLAG标签的重要供应商MilliporeSigma。
表10 蛋白质纯化技术引用调查统计。(num:发布的数量)
| 纯化类型 | 供应商 | 主要品牌 | num | 参考文献 |
| Affinity(亲和纯化) | ||||
| HIS | Qiagen | Ni-NTA agarose/resin | 61 | [59, 60] |
| GE Healthcare | Ni Sepharose, HisTrap FF/HP | 35 | [60, 61] | |
| Clontech | TALON metal affinity | 22 | [62, 63] | |
| Thermo Fisher / Invitrogen | HisPur Cobalt/Ni-NTA resin | 8 | [64, 65] | |
| GST | GE Healthcare | glutathione Sepharose 4B | 64 | [63, 66] |
| MilliporeSigma | glutathione agarose | 3 | [67, 68] | |
| FLAG | MilliporeSigma | FLAG M2 affinity | 17 | [64, 69] |
| DNA binding protein | GE Healthcare | heparin | 15 | [70, 71] |
| maltose-binding protein | New England Biolabs | amylose resin | 8 | [71, 72] |
| Other cited affinity-based protein purification systems include Vector Laboratories agarose-bound Jacalin for 0-linked glycoproteins [73], strepbiotin-based systems from Thermo Fisher Pierce [74, 75], from MilliporeSigma [76], from Qiagen [77], from IBA [78], and from MilliporeSigma [74], MilliporeSigma Myc immunoaffinity resin [79], GE Healthcare poly(A)-Sepharose 4B [14], Thermo Scientific SulfoLink resin [80], BioRad Affi-Gel 10 resin [80], RepliGen protein A agarose [80] Bio-Rad Affigel [80, 81]. | ||||
| size exclusion(尺寸排阻层析法) | ||||
| GE-Healthcare | Superdex / Superose / Sephacryl | 96 | [59, 82] | |
| ion exchange(离子交换层析) | ||||
| anion | GE Healthcare | Mono Q column | 10 | [59, 64] |
| GE Healthcare | HiTrap Q | 6 | [83, 84] | |
| GE Healthcare | Source 15Q | 3 | [59, 67] | |
| GE Healthcare | Q-Sepharose | 1 | [85] | |
| Whatman | DE52 anion exchange resin | 1 | [86] | |
| MilliporeSigma | DE52 anion exchange column | 1 | [61] | |
| cation | GE Healthcare | Mono S column | 6 | [15, 87] |
| TOROH | TSKgelSP-5PW column | 1 | [59] | |
| GE Healthcare | Source 15S | 1 | [59] | |
| hydrophobic interaction(疏水作用层析法) | ||||
| GE healthcare | phenyl sepharose CL-4B | 1 | [88] | |
| MilliporeSigma | phenyl sepharose | 1 | [85] | |
在调查的文献中,通过millipoma BugBuster[89, 90]或AVESTIN flex C-3 cell disruptor[91, 92]制备蛋白样品,通过millipoma Amicon Ultra concentrators[60, 93]、Thermo Fisher Sartorius spin columns[94]或Vivaspin concentrators[70, 95, 96]进行浓缩。Maini Rekdal V等人通过Avestin emulsiflex C3 细胞干扰素、Thermo Fisher Scientific HisPur Ni-NTA树脂和VMR自旋柱(目录号97027-9)从大肠杆菌BL21(DE3)中纯化了E. faecalis MMH594酪氨酸脱羧酶[65]。
References
- Good N, Winget G, Winter W, Connolly T, Izawa S, Singh R. Hydrogen ion buffers for biological research. Biochemistry. 1966;5:467-77 pubmed
- Grady J, Chasteen N, Harris D. Radicals from "Good's" buffers. Anal Biochem. 1988;173:111-5 pubmed
- Desmarais W, Bienvenue D, Bzymek K, Holz R, Petsko G, Ringe D. The 1.20 A resolution crystal structure of the aminopeptidase from Aeromonas proteolytica complexed with tris: a tale of buffer inhibition. Structure. 2002;10:1063-72 pubmed
- Ghalanbor Z, Ghaemi N, Marashi S, Amanlou M, Habibi Rezaei M, Khajeh K, et al. Binding of Tris to Bacillus licheniformis alpha-amylase can affect its starch hydrolysis activity. Protein Pept Lett. 2008;15:212-4 pubmed
- Burgess R. Refolding solubilized inclusion body proteins. Methods Enzymol. 2009;463:259-82 pubmedpublisher
- Singh A, Upadhyay V, Upadhyay A, Singh S, Panda A. Protein recovery from inclusion bodies of Escherichia coli using mild solubilization process. Microb Cell Fact. 2015;14:41 pubmedpublisher
- Scott M, Modha S, Rhodes A, Broadway N, Hardwicke P, Zhao H, et al. Efficient expression of secreted proteases via recombinant BacMam virus. Protein Expr Purif. 2007;52:104-16 pubmed
- Grabski A. Advances in preparation of biological extracts for protein purification. Methods Enzymol. 2009;463:285-303 pubmedpublisher
- Pejchal R, Doores K, Walker L, Khayat R, Huang P, Wang S, et al. A potent and broad neutralizing antibody recognizes and penetrates the HIV glycan shield. Science. 2011;334:1097-103 pubmedpublisher
- Cruz Migoni A, Hautbergue G, Artymiuk P, Baker P, Bokori Brown M, Chang C, et al. A Burkholderia pseudomallei toxin inhibits helicase activity of translation factor eIF4A. Science. 2011;334:821-4 pubmedpublisher
- Shenoy A, Wellington D, Kumar P, Kassa H, Booth C, Cresswell P, et al. GBP5 promotes NLRP3 inflammasome assembly and immunity in mammals. Science. 2012;336:481-5 pubmedpublisher
- Hernandez J, Stein A, Behrmann E, Riedel D, Cypionka A, Farsi Z, et al. Membrane fusion intermediates via directional and full assembly of the SNARE complex. Science. 2012;336:1581-4 pubmedpublisher
- Mukherjee S, Behar M, Birnbaum H, Hoffmann A, Wright P, Ghosh G. Analysis of the RelA:CBP/p300 interaction reveals its involvement in NF-?B-driven transcription. PLoS Biol. 2013;11:e1001647 pubmedpublisher
- Bohne A, Schwarz C, Schottkowski M, Lidschreiber M, Piotrowski M, Zerges W, et al. Reciprocal regulation of protein synthesis and carbon metabolism for thylakoid membrane biogenesis. PLoS Biol. 2013;11:e1001482 pubmedpublisher
- Lai Y, Diao J, Cipriano D, Zhang Y, Pfuetzner R, Padolina M, et al. Complexin inhibits spontaneous release and synchronizes Ca2+-triggered synaptic vesicle fusion by distinct mechanisms. elife. 2014;3:e03756 pubmedpublisher
- Hervás R, Oroz J, Galera Prat A, Goñi O, Valbuena A, Vera A, et al. Common features at the start of the neurodegeneration cascade. PLoS Biol. 2012;10:e1001335 pubmedpublisher
- Schroeder C, Ostrem J, Hertz N, Vale R. A Ras-like domain in the light intermediate chain bridges the dynein motor to a cargo-binding region. elife. 2014;3:e03351 pubmedpublisher
- Stokes J, Davis J, Mangat C, Williamson J, Brown E. Discovery of a small molecule that inhibits bacterial ribosome biogenesis. elife. 2014;3:e03574 pubmedpublisher
- Arakawa T, Philo J, Tsumoto K, Yumioka R, Ejima D. Elution of antibodies from a Protein-A column by aqueous arginine solutions. Protein Expr Purif. 2004;36:244-8 pubmed
- Chong S, Mersha F, Comb D, Scott M, Landry D, Vence L, et al. Single-column purification of free recombinant proteins using a self-cleavable affinity tag derived from a protein splicing element. Gene. 1997;192:271-81 pubmed
- Jochum C, Beste M, Stone D, Graves S, Storb R. Development and in vitro characterization of canine CD40-Ig. Vet Immunol Immunopathol. 2008;123:260-5 pubmedpublisher
- Corti D, Voss J, Gamblin S, Codoni G, Macagno A, Jarrossay D, et al. A neutralizing antibody selected from plasma cells that binds to group 1 and group 2 influenza A hemagglutinins. Science. 2011;333:850-6 pubmedpublisher
- Shen Y, Tenney A, Busch S, Horn K, Cuascut F, Liu K, et al. PTPsigma is a receptor for chondroitin sulfate proteoglycan, an inhibitor of neural regeneration. Science. 2009;326:592-6 pubmedpublisher
- Li F, Ravetch J. Inhibitory Fc? receptor engagement drives adjuvant and anti-tumor activities of agonistic CD40 antibodies. Science. 2011;333:1030-4 pubmedpublisher
- Wichmann A, Borg H. Purification of human immunoglobulin M by affinity chromatography on protamine-Sepharose. Biochim Biophys Acta. 1977;490:363-9 pubmed
- Salaman M, Williamson A. Isoelectric focusing of proteins in the native and denatured states. Anomalous behaviour of plasma albumin. Biochem J. 1971;122:93-9 pubmed
- Vesterberg O. Isoelectric fractionation, analysis, and characterization of ampholytes in natural pH gradients. V. Separation of myoglobins and studies on their electro-chemical differences. Acta Chem Scand. 1967;21:206-16 pubmed
- Awdeh Z, Williamson A, Askonas B. Isoelectric focusing in polyacrylamide gel and its application to immunoglobulins. Nature. 1968;219:66-7 pubmed
- Righetti P. Determination of the isoelectric point of proteins by capillary isoelectric focusing. J Chromatogr A. 2004;1037:491-9 pubmed
- Pihlasalo S, Auranen L, Hanninen P, Harma H. Method for estimation of protein isoelectric point. Anal Chem. 2012;84:8253-8 pubmedpublisher
- Ahamed T, Chilamkurthi S, Nfor B, Verhaert P, van Dedem G, van der Wielen L, et al. Selection of pH-related parameters in ion-exchange chromatography using pH-gradient operations. J Chromatogr A. 2008;1194:22-9 pubmed
- Trodler P, Nieveler J, Rusnak M, Schmid R, Pleiss J. Rational design of a new one-step purification strategy for Candida antarctica lipase B by ion-exchange chromatography. J Chromatogr A. 2008;1179:161-7 pubmed
- Duncan J, Chen A, Siebert C. Performance evaluation of non-porous versus porous ion-exchange packings in the separation of proteins by high-performance liquid chromatography. J Chromatogr. 1987;397:3-12 pubmed
- Staby A, Jensen R, Bensch M, Hubbuch J, Dünweber D, Krarup J, et al. Comparison of chromatographic ion-exchange resins VI. Weak anion-exchange resins. J Chromatogr A. 2007;1164:82-94 pubmed
- DePhillips P, Lenhoff A. Determinants of protein retention characteristics on cation-exchange adsorbents. J Chromatogr A. 2001;933:57-72 pubmed
- von Hippel P, Wong K. On the conformational stability of globular proteins. The effects of various electrolytes and nonelectrolytes on the thermal ribonuclease transition. J Biol Chem. 1965;240:3909-23 pubmed
- Tsumoto K, Ejima D, Senczuk A, Kita Y, Arakawa T. Effects of salts on protein-surface interactions: applications for column chromatography. J Pharm Sci. 2007;96:1677-90 pubmed
- Er el Z, Zaidenzaig Y, Shaltiel S. Hydrocarbon-coated sepharoses. Use in the purification of glycogen phosphorylase. Biochem Biophys Res Commun. 1972;49:383-90 pubmed
- Woodbury R, Hardy S, Randall L. Complex behavior in solution of homodimeric SecA. Protein Sci. 2002;11:875-82 pubmed
- Corbett R, Roche R. Use of high-speed size-exclusion chromatography for the study of protein folding and stability. Biochemistry. 1984;23:1888-94 pubmed
- Kim T, Paik S, Yang C. Structural and functional implications of C-terminal regions of alpha-synuclein. Biochemistry. 2002;41:13782-90 pubmed
- Hjerten S, Levin O, Tiselius A. Protein chromatography on calcium phosphate columns. Arch Biochem Biophys. 1956;65:132-55 pubmed
- Cummings L, Snyder M, Brisack K. Protein chromatography on hydroxyapatite columns. Methods Enzymol. 2009;463:387-404 pubmedpublisher
- Hilbrig F, Freitag R. Isolation and purification of recombinant proteins, antibodies and plasmid DNA with hydroxyapatite chromatography. Biotechnol J. 2012;7:90-102 pubmedpublisher
- Giri L. Chromatofocusing. Methods Enzymol. 1990;182:380-92 pubmed
- Schenkman J, Jansson I. Spectral analyses of cytochromes P450. Methods Mol Biol. 2006;320:11-8 pubmed
- Koehn J, Hunt I. High-Throughput Protein Production (HTPP): a review of enabling technologies to expedite protein production. Methods Mol Biol. 2009;498:1-18 pubmedpublisher
- Kim Y, Babnigg G, Jedrzejczak R, Eschenfeldt W, Li H, Maltseva N, et al. High-throughput protein purification and quality assessment for crystallization. Methods. 2011;55:12-28 pubmedpublisher
- SGC | Integral Membrane Proteins. Available from: thesgc.org/science/imp
- Lin S, Guidotti G. Purification of membrane proteins. Methods Enzymol. 2009;463:619-29 pubmedpublisher
- Duquesne K, Sturgis J. Membrane protein solubilization. Methods Mol Biol. 2010;601:205-17 pubmedpublisher
- Roy A. Membrane preparation and solubilization. Methods Enzymol. 2015;557:45-56 pubmedpublisher
- Linke D. Detergents: an overview. Methods Enzymol. 2009;463:603-17 pubmedpublisher
- Arachea B, Sun Z, Potente N, Malik R, Isailovic D, Viola R. Detergent selection for enhanced extraction of membrane proteins. Protein Expr Purif. 2012;86:12-20 pubmedpublisher
- He Y, Wang K, Yan N. The recombinant expression systems for structure determination of eukaryotic membrane proteins. Protein Cell. 2014;5:658-72 pubmedpublisher
- Anandan A, Vrielink A. Detergents in Membrane Protein Purification and Crystallisation. Adv Exp Med Biol. 2016;922:13-28 pubmedpublisher
- Smith S. Strategies for the Purification of Membrane Proteins. Methods Mol Biol. 2017;1485:389-400 pubmed
- Mitra N, Liu Y, Liu J, Serebryany E, Mooney V, DeVree B, et al. Calcium-dependent ligand binding and G-protein signaling of family B GPCR parathyroid hormone 1 receptor purified in nanodiscs. ACS Chem Biol. 2013;8:617-25 pubmedpublisher
- Gibson B, Doolittle L, Schneider M, Jensen L, Gamarra N, Henry L, et al. Organization of Chromatin by Intrinsic and Regulated Phase Separation. Cell. 2019;179:470-484.e21 pubmedpublisher
- Zhao N, Kamijo K, Fox P, Oda H, Morisaki T, Sato Y, et al. A genetically encoded probe for imaging nascent and mature HA-tagged proteins in vivo. Nat Commun. 2019;10:2947 pubmedpublisher
- Pastushok L, Fu Y, Lin L, Luo Y, DeCoteau J, Lee K, et al. A Novel Cell-Penetrating Antibody Fragment Inhibits the DNA Repair Protein RAD51. Sci Rep. 2019;9:11227 pubmedpublisher
- Ślężak T, Bailey L, Jaskolowski M, Nahotko D, Filippova E, Davydova E, et al. An engineered ultra-high affinity Fab-Protein G pair enables a modular antibody platform with multifunctional capability. Protein Sci. 2019;: pubmedpublisher
- Bhogaraju S, Bonn F, Mukherjee R, Adams M, Pfleiderer M, Galej W, et al. Inhibition of bacterial ubiquitin ligases by SidJ-calmodulin catalysed glutamylation. Nature. 2019;572:382-386 pubmedpublisher
- Kuper J, Braun C, Elias A, Michels G, Sauer F, Schmitt D, et al. In TFIIH, XPD helicase is exclusively devoted to DNA repair. PLoS Biol. 2014;12:e1001954 pubmedpublisher
- Maini Rekdal V, Bess E, Bisanz J, Turnbaugh P, Balskus E. Discovery and inhibition of an interspecies gut bacterial pathway for Levodopa metabolism. Science. 2019;364: pubmedpublisher
- Polley S, Huang D, Hauenstein A, Fusco A, Zhong X, Vu D, et al. A structural basis for I?B kinase 2 activation via oligomerization-dependent trans auto-phosphorylation. PLoS Biol. 2013;11:e1001581 pubmedpublisher
- Zalatan J, Coyle S, Rajan S, Sidhu S, Lim W. Conformational control of the Ste5 scaffold protein insulates against MAP kinase misactivation. Science. 2012;337:1218-22 pubmedpublisher
- Johnson K, Zhu S, Tremblay M, Payette J, Wang J, Bouchez L, et al. A stem cell-based approach to cartilage repair. Science. 2012;336:717-21 pubmedpublisher
- Puel A, Cypowyj S, Bustamante J, Wright J, Liu L, Lim H, et al. Chronic mucocutaneous candidiasis in humans with inborn errors of interleukin-17 immunity. Science. 2011;332:65-8 pubmedpublisher
- Hsiao Y, Fang W, Lee C, Chen Y, Yuan H. Structural insights into DNA repair by RNase T--an exonuclease processing 3' end of structured DNA in repair pathways. PLoS Biol. 2014;12:e1001803 pubmedpublisher
- Lamech L, Mallam A, Lambowitz A. Evolution of RNA-protein interactions: non-specific binding led to RNA splicing activity of fungal mitochondrial tyrosyl-tRNA synthetases. PLoS Biol. 2014;12:e1002028 pubmedpublisher
- Pan C, Potratz J, Cannon B, Simpson Z, Ziehr J, Tijerina P, et al. DEAD-box helicase proteins disrupt RNA tertiary structure through helix capture. PLoS Biol. 2014;12:e1001981 pubmedpublisher
- Inamori K, Yoshida Moriguchi T, Hara Y, Anderson M, Yu L, Campbell K. Dystroglycan function requires xylosyl- and glucuronyltransferase activities of LARGE. Science. 2012;335:93-6 pubmedpublisher
- Seth D, Hausladen A, Wang Y, Stamler J. Endogenous protein S-Nitrosylation in E. coli: regulation by OxyR. Science. 2012;336:470-3 pubmedpublisher
- Zhang M, Wu P, Kelly F, Nurse P, Hang H. Quantitative control of protein S-palmitoylation regulates meiotic entry in fission yeast. PLoS Biol. 2013;11:e1001597 pubmedpublisher
- Previs M, Beck Previs S, Gulick J, Robbins J, Warshaw D. Molecular mechanics of cardiac myosin-binding protein C in native thick filaments. Science. 2012;337:1215-8 pubmedpublisher
- Lazarus J, Moughamian A, Tokito M, Holzbaur E. Dynactin subunit p150(Glued) is a neuron-specific anti-catastrophe factor. PLoS Biol. 2013;11:e1001611 pubmedpublisher
- Li Z, Park Y, Marcotte E. A Bacteriophage tailspike domain promotes self-cleavage of a human membrane-bound transcription factor, the myelin regulatory factor MYRF. PLoS Biol. 2013;11:e1001624 pubmedpublisher
- Yi W, Clark P, Mason D, Keenan M, Hill C, Goddard W, et al. Phosphofructokinase 1 glycosylation regulates cell growth and metabolism. Science. 2012;337:975-80 pubmedpublisher
- Farcas A, Blackledge N, Sudbery I, Long H, McGouran J, Rose N, et al. KDM2B links the Polycomb Repressive Complex 1 (PRC1) to recognition of CpG islands. elife. 2012;1:e00205 pubmedpublisher
- Zhou H, Kaplan T, Li Y, Grubisic I, Zhang Z, Wang P, et al. Dual functions of TAF7L in adipocyte differentiation. elife. 2013;2:e00170 pubmedpublisher
- Maun H, Jackman J, Choy D, Loyet K, Staton T, Jia G, et al. An Allosteric Anti-tryptase Antibody for the Treatment of Mast Cell-Mediated Severe Asthma. Cell. 2019;179:417-431.e19 pubmedpublisher
- Schulz S, Iglesias Cans M, Krah A, Yildiz O, Leone V, Matthies D, et al. A new type of Na(+)-driven ATP synthase membrane rotor with a two-carboxylate ion-coupling motif. PLoS Biol. 2013;11:e1001596 pubmedpublisher
- Walser R, Burke J, Gogvadze E, Bohnacker T, Zhang X, Hess D, et al. PKC? phosphorylates PI3K? to activate it and release it from GPCR control. PLoS Biol. 2013;11:e1001587 pubmedpublisher
- Diao J, Burré J, Vivona S, Cipriano D, Sharma M, Kyoung M, et al. Native α-synuclein induces clustering of synaptic-vesicle mimics via binding to phospholipids and synaptobrevin-2/VAMP2. elife. 2013;2:e00592 pubmedpublisher
- Pinheiro V, Taylor A, Cozens C, Abramov M, Renders M, Zhang S, et al. Synthetic genetic polymers capable of heredity and evolution. Science. 2012;336:341-4 pubmedpublisher
- Vemu A, Szczęsna E, Zehr E, Spector J, Grigorieff N, Deaconescu A, et al. Severing enzymes amplify microtubule arrays through lattice GTP-tubulin incorporation. Science. 2018;361: pubmedpublisher
- Okada C, Yamashita E, Lee S, Shibata S, Katahira J, Nakagawa A, et al. A high-resolution structure of the pre-microRNA nuclear export machinery. Science. 2009;326:1275-9 pubmedpublisher
- Azoitei M, Correia B, Ban Y, Carrico C, Kalyuzhniy O, Chen L, et al. Computation-guided backbone grafting of a discontinuous motif onto a protein scaffold. Science. 2011;334:373-6 pubmedpublisher
- Zhang S, Bryant D. The tricarboxylic acid cycle in cyanobacteria. Science. 2011;334:1551-3 pubmedpublisher
- Fleishman S, Whitehead T, Ekiert D, Dreyfus C, Corn J, Strauch E, et al. Computational design of proteins targeting the conserved stem region of influenza hemagglutinin. Science. 2011;332:816-21 pubmedpublisher
- Gardner B, Walter P. Unfolded proteins are Ire1-activating ligands that directly induce the unfolded protein response. Science. 2011;333:1891-4 pubmedpublisher
- Rice H, de Malmazet D, Schreurs A, Frere S, Van Molle I, Volkov A, et al. Secreted amyloid-β precursor protein functions as a GABABR1a ligand to modulate synaptic transmission. Science. 2019;363: pubmedpublisher
- McNulty N, Wu M, Erickson A, Pan C, Erickson B, Martens E, et al. Effects of diet on resource utilization by a model human gut microbiota containing Bacteroides cellulosilyticus WH2, a symbiont with an extensive glycobiome. PLoS Biol. 2013;11:e1001637 pubmedpublisher
- Mechaly A, Sassoon N, Betton J, Alzari P. Segmental helical motions and dynamical asymmetry modulate histidine kinase autophosphorylation. PLoS Biol. 2014;12:e1001776 pubmedpublisher
- Fleckenstein M, Reese M, Könen Waisman S, Boothroyd J, Howard J, Steinfeldt T. A Toxoplasma gondii pseudokinase inhibits host IRG resistance proteins. PLoS Biol. 2012;10:e1001358 pubmedpublisher
ISSN : 2329-5139



